-
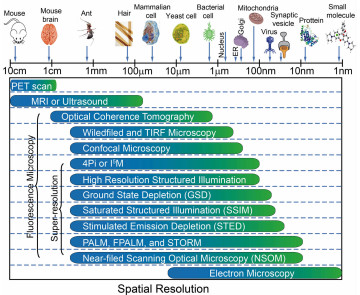
摘要: 为了进一步认知复杂环境中的细胞生物学过程,研究人员发展了各种各样的生物成像技术。在这些技术中,生物荧光成像因简单的成像条件以及对生物样品的相容性而得到了广泛的发展。然而,传统的荧光成像技术受到了光学衍射极限的限制,无法分辨低于200 nm的空间结构,阻碍了对亚细胞结构的生物学过程研究。超分辨荧光显微镜技术突破了传统光学衍射对成像分辨率的限制,能够获取纳米尺度的细胞动态过程。除了对传统的宽场荧光显微镜框架的改进及升级改造之外,目前典型的超分辨成像显微镜技术通常依赖于荧光探针材料的光物理性质。常用的荧光探针材料包括荧光蛋白、有机荧光分子和纳米荧光材料等。本文介绍了几种主流的超分辨荧光显微成像技术并总结了已经成功应用到超分辨生物荧光成像中的荧光探针材料的应用进展。Abstract: In order to further understand the biological cellular processes in the complex environments, a variety of bioimaging techniques have been developed by researchers. Biofluorescence imaging has been extensively developed due to its simple imaging conditions and compatibility with biological samples. However, the traditional fluorescence imaging technology is restricted by the optical diffraction limit, so it is impossible to resolve the spatial structure below 200 nm, which hinders the study of the biological processes of subcellular structures. Super-resolution fluorescence microscopy breaks through the limitations of imaging resolution with traditional optical diffraction and can acquire nanoscale cellular dynamics. In addition to improvements and upgrades to traditional wide-field fluorescence microscope frames, typical super-resolution imaging microscopy techniques currently also rely on the photophysical properties of fluorescent probe materials. Commonly used fluorescent probe materials mainly include fluorescent proteins, organic fluorescent molecules and fluorescent nanomaterials. This paper introduces several mainstream super-resolution fluorescence microscopy techniques and summarizes the application status of fluorescent probe materials that have been successfully applied to super-resolution biofluorescence imaging.
-
图 7 光激活定位显微镜和随机光学重构显微镜工作原理及亚细胞结构超分辨图(左侧:PALM及COS-7细胞溶酶体超分辨图像;右侧:STORM原理图及亚细胞结构单色、多色超分辨图像)[14, 17]
Figure 7. Working principle of Photoactivation Localization Microscopy, Stochastic Optical Reconstruction Microscopy and subcellular structure super resolution images(Left: PALM and COS-7 cell lysosomal superresolution images; right:STORM schematic and subcellular structure monochrome, multicolor super-resolution images)[14, 17]
图 12 (a~c)3种不同DNA结构的三色STORM图像(Alexa405/Cy5, Cy2/Cy5和Cy3/Cy5标记;405 nm, 457 nm和532 nm激光激发);(d)Alexa647/Cy3标记的BC-S-1细胞内网格蛋白凹坑(CCPs)的三维STORM图像[54-55]
Figure 12. (a~c)Three-color STORM images of three different DNA constructs(labelled with Alexa405/Cy5, Cy2/Cy5, and Cy3/Cy5, activated using 405, 457 and 532 nm laser, respectively); (d)3D STORM image of clathrin-coated pits(CCPs) stained with the Alexa647/Cy3 in BS-C-1 cells[54-55]
图 14 STORM荧光探针41~48结构式及细胞微管的超分辨图像[62];Si-罗丹明荧光分子(49~55)衍生物以及分子54在合适pH环境中闪烁机制的模型及相应的化学结构式[65]; ATTO655标记的CCR5在水和重水不同环境下,分子56和57在光照时互相转化过程以及亚细胞结构图像[66]
Figure 14. Chemical structures of fluorescent dyes(41-48) used in STORM[62]; Chemical structures of 49-55 and proposed photoblinking mechanism of 54 in a suitable pH environment[65]; Photoswitching between 56 and 57 upon photoirradiation in the presence of oxygen and ATTO655-labelled CCR5 imaging of live cells in H2O and D2O[66]
-
[1] FERRARI M. Cancer nanotechnology:opportunities and challenges[J]. Nature Reviews:Cancer, 2005, 5(3):161-171. doi: 10.1038/nrc1566 [2] NIE S, XING Y, KIM G J, et al.. Nanotechnology applications in cancer[J]. Annual Review of Biomedical Engineering, 2007, 9:257-288. doi: 10.1146/annurev.bioeng.9.060906.152025 [3] BEN N G GIEPMANS, STEPHEN R ADAMS, MARK H ELLISMAN, et al.. The fluorescent toolbox for assessing protein location and function[J]. Science, 2006, 312(217):224. http://cn.bing.com/academic/profile?id=cc996e1880426cdd60a036f11b646509&encoded=0&v=paper_preview&mkt=zh-cn [4] LI G W, XIE X S. Central dogma at the single-molecule level in living cells[J]. Nature, 2011, 475(7356):308-315. doi: 10.1038/nature10315 [5] XIE X. S, YU J, YANG W Y. Living cells as test tubes[J]. Science, 2006, 312:228-230. doi: 10.1126/science.1127566 [6] ABBE E. Beiträge zur Theorie des Mikroskops und der mikroskopischen Wahrnehmung[J]. Archiv Für Mikroskopische Anatomie, 1873, 9(1):413-418. doi: 10.1007/BF02956173 [7] KLEIN T, PROPPERT S, SAUER M. Eight years of single-molecule localization microscopy[J]. Histochemistry and Cell Biology, 2014, 141(6):561-575. doi: 10.1007/s00418-014-1184-3 [8] STEFAN W. H, WICHMANN J. Breaking the diffraction resolution limit by stimulated emission stimulatedemission depletion fluorescence microscopy[J]. Optics Letters, 1994, 19(11):780-782. doi: 10.1364/OL.19.000780 [9] GAEL M, REBECCA M, BIRKA H, et al.. Fast STED microscopy with continuous wave fiber lasers[J]. Optics Express, 2010, 18(2):1302-1309. doi: 10.1364/OE.18.001302 [10] SUSANNE S, THORSTEN S, RITTWEGER E, et al.. STED nanoscopy with mass-produced laser diodes[J]. Optics Express, 2011, 19(9):8066-8072. doi: 10.1364/OE.19.008066 [11] ROUBINET B, MARIANO L. B, PHILIPP A, et al.. Carboxylated photoswitchable diarylethenes for biolabeling and super-resolution RESOLFT microscopy[J]. Angew Chem. Int. Ed. Engl., 2016, 55:15429-15433. doi: 10.1002/anie.v55.49 [12] BOHM U, HELL S W, SCHMIDT R. 4Pi-RESOLFT nanoscopy[J]. Nat. Commun., 2016, 7:10504. doi: 10.1038/ncomms10504 [13] HOFMANN M, EGGELING C, JAKOBS S, et al.. Breaking the diffraction barrier in fluorescence microscopy at low light intensities by using reversibly photoswitchable proteins[J]. Proc. Natl. Acad. Sci. USA, 2005, 102(49):17565-17569. doi: 10.1073/pnas.0506010102 [14] BETZIG E, PATTERSON G H, SOUGRAT R, et al.. Imaging intracellular fluorescent proteins at nanometer resolution[J]. Science, 2006, 313(5793):1642-1645. doi: 10.1126/science.1127344 [15] LEGANTW R, SHAO L, GRIMM J B, et al.. High-density three-dimensional localization microscopy across large volumes[J]. Nature Methods, 2016, 13:359-365. doi: 10.1038/nmeth.3797 [16] EN CAI, KYLE MARCHUK, PETER BEEMILLER, et al.. Visualizing dynamic microvillar search and stabilization during ligand detection by T cells[J]. Science, 2017, 356:598. http://cn.bing.com/academic/profile?id=3a71d2c194dd6a935895951c68f2e752&encoded=0&v=paper_preview&mkt=zh-cn [17] RUST M J, BATES M, ZHUANG X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy(STORM)[J]. Nat Methods, 2006, 3(10):793-795. doi: 10.1038/nmeth929 [18] VAN D L S, LOSCHBERGER A, KLEIN T, et al.. Direct stochastic optical reconstruction microscopy with standard fluorescent probes[J]. Nat. Protoc., 2011, 6(7):991-1009. doi: 10.1038/nprot.2011.336 [19] DERTINGER T, COLYER R, IYER G, et al.. Fast, background-free, 3D super-resolution optical fluctuation imaging(SOFI)[J]. Proc. Natl. Acad. Sci. USA, 2009, 106(52):22287-22292. doi: 10.1073/pnas.0907866106 [20] COX S, ROSTEN E, MONYPENNY J, et al.. Bayesian localization microscopy reveals nanoscale podosome dynamics[J]. Nat. Methods, 2011, 9(2):195-200. http://cn.bing.com/academic/profile?id=c07b5ae46a81588374ddd70390807f47&encoded=0&v=paper_preview&mkt=zh-cn [21] CHEN X Z, WEI M, ZHENG M M, et al.. Study of RNA polymerase Ⅱ clustering inside live-cell nuclei using bayesian nanoscopy[J]. ACS Nano, 2016, 10(2):2447-2454. doi: 10.1021/acsnano.5b07257 [22] GUSTAFSSON M G L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy[J]. Journal of Microscopy, 2000, 192:82-87. http://cn.bing.com/academic/profile?id=4a44fafc541473968a8d9f3d53236a52&encoded=0&v=paper_preview&mkt=zh-cn [23] GUSTAFSSON M G. Nonlinear structured-illumination microscopy:wide-field fluorescence imaging with theoretically unlimited resolution[J]. Proc. Natl. Acad. Sci. USA, 2005, 102(37):13081-13086. doi: 10.1073/pnas.0406877102 [24] HELL T A K S W. Subdiffraction resolution in far-field fluorescence microscopy[J]. Optics Letters, 1999, 24(14):954-956. doi: 10.1364/OL.24.000954 [25] HEIN B, WILLIG K I, HELL S W. Stimulated emission depletion(STED) nanoscopy of a fluorescent protein-labeled organelle inside a living cell[J]. Proc. Natl. Acad. Sci. USA, 2008, 105(38):14271-14276. doi: 10.1073/pnas.0807705105 [26] ALEXEY N BUTKEVICH, GYUZEL YU MITRONOVA, SVEN C SIDENSTEIN, et al.. Fluorescent rhodamines and fluorogenic carbopyronines for super-resolution STED microscopy in living cells[J]. Angew Chem. Int. Ed. Engl., 2016, 55:3290-3294. doi: 10.1002/anie.201511018 [27] BORDENAVE M D, BALZAROTTI F, STEFANI F D, et al.. STED nanoscopy with wavelengths at the emission maximum[J]. Journal of Physics D:Applied Physics, 2016, 49(36):365102. doi: 10.1088/0022-3727/49/36/365102 [28] GOTTFERT F, PLEINER T, HEINE J, et al.. Strong signal increase in STED fluorescence microscopy by imaging regions of subdiffraction extent[J]. Proc. Natl. Acad. Sci. USA, 2017, 114(9):2125-2130. doi: 10.1073/pnas.1621495114 [29] HELL S W, KROUG M. Ground-state-depletion fluorscence microscopy:a concept for breaking the diffraction resolution limit[J]. Applied Physics B, 1995, 60(5):495-497. doi: 10.1007/BF01081333 [30] JOANNA O, ADOLFSSON K, WESTPHAL V, et al.. Ground state depletion nanoscopy resolves semiconductor nanowire barcode segments at room temperature[J]. Nano Letters, 2017, 17(4):2652-2659. doi: 10.1021/acs.nanolett.7b00468 [31] WURM C A, KOLMAKOV K, GÖTTFERT F, et al.. Novel red fluorophores with superior performance in STED microscopy[J]. Optical Nanoscopy, 2012, 1(1):7. doi: 10.1186/2192-2853-1-7 [32] SCHILL H, NIZAMOV S, BOTTANELLI F, et al.. 4-Trifluoromethyl-substituted coumarins with large Stokes shifts:synthesis, bioconjugates, and their use in super-resolution fluorescence microscopy[J]. Chemistry, 2013, 19(49):16556-16565. doi: 10.1002/chem.201302037 [33] ERDMANN R S, TAKAKURA H, THOMPSON A D, et al.. Super-resolution imaging of the Golgi in live cells with a bioorthogonal ceramide probe[J]. Angew Chem. Int. Ed. Engl., 2014, 53(38):10242-10246. doi: 10.1002/anie.201403349 [34] LUKINAVICIUS G, REYMOND L, D'ESTE E, et al.. Fluorogenic probes for live-cell imaging of the cytoskeleton[J]. Nat. Methods, 2014, 11(7):731-733. doi: 10.1038/nmeth.2972 [35] D'ESTE E, KAMIN D, GOTTFERT F, et al.. STED nanoscopy reveals the ubiquity of subcortical cytoskeleton periodicity in living neurons[J]. Cell Rep., 2015, 10(8):1246-1251. doi: 10.1016/j.celrep.2015.02.007 [36] KOLMAKOV K, HEBISCH E, WOLFRAM T, et al.. Far-red emitting fluorescent dyes for optical nanoscopy:fluorinated Silicon-Rhodamines(SiRF Dyes) and phosphorylated oxazines[J]. Chemistry, 2015, 21(38):13344-13356. doi: 10.1002/chem.201501394 [37] KASPER R, HARKE B, FORTHMANN C, et al.. Single-molecule STED microscopy with photostable organic fluorophores[J]. Small, 2010, 6(13):1379-1384. doi: 10.1002/smll.v6:13 [38] GRAZVYDAS L, REYMOND L, UMEZAWA K, et al.. Fluorogenic probes for multicolor imaging in living cells[J]. J. Am. Chem. Soc., 2016, 138:9365-9368. doi: 10.1021/jacs.6b04782 [39] HANNE J, FALK H J, GORLITZ F, et al.. STED nanoscopy with fluorescent quantum dots[J]. Nat. Commun., 2015, 6:7127. doi: 10.1038/ncomms8127 [40] AHMET YILDIZ, JOSEPH N FORKEY, SEAN A MCKINNEY, et al.. Myosin V walks hand-over-hand_single fluorophore imaging with 1.5-nm localization[J]. Science, 2003, 300(27):2061-2065. http://cn.bing.com/academic/profile?id=66c301b8c26d4db38977901e170978a0&encoded=0&v=paper_preview&mkt=zh-cn [41] ALISTAIR N. BOETTIGER, BOGDAN BINTU, JEFFREY R. MOFFITT, et al.. Super-resolution imaging reveals distinct chromatin folding for different epigenetic states[J]. Nature, 2016. http://cn.bing.com/academic/profile?id=e46d0bcf905784cbe271fb4dcded58cf&encoded=0&v=paper_preview&mkt=zh-cn [42] BELIVEAU B J, BOETTIGER A N, AVENDANO M S, et al.. Single-molecule super-resolution imaging of chromosomes and in situ haplotype visualization using Oligopaint FISH probes[J]. Nat. Commun., 2015, 6:7147. doi: 10.1038/ncomms8147 [43] VISWANATHAN S, WILLIAMS M E, BLOSS E B, et al.. High-performance probes for light and electron microscopy[J]. Nat. Methods, 2015, 12(6):568-576. doi: 10.1038/nmeth.3365 [44] ZHANG X, ZHANG M S, DONG L, et al.. Highly photostable, reversibly photoswitchable fluorescent protein with high contrast ratio for live-cell superresolution microscopy[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016. http://cn.bing.com/academic/profile?id=5658cff492d84e0cac790d52988931b2&encoded=0&v=paper_preview&mkt=zh-cn [45] LEE H-L D, LORD S J, SHIGEKI I, et al.. Superresolution imaging of targeted proteins in fixed and living cells using photoactivatable organic fluorophores[J]. J. Am. Chem. Soc., 2010, 132:15099-15101. doi: 10.1021/ja1044192 [46] LEE M K, WILLIAMS J, TWIEG R J, et al.. Enzymatic activation of nitro-aryl fluorogens in live bacterial cells for enzymatic turnover-activated localization microscopydagger[J]. Chemical Science, 2013, 42:220-225. http://cn.bing.com/academic/profile?id=a58f8be5b6a9afd90fc52e7c1d64f0d2&encoded=0&v=paper_preview&mkt=zh-cn [47] FOLLING J, BELOV V, KUNETSKY R, et al.. Photochromic rhodamines provide nanoscopy with optical sectioning[J]. Angew. Chem. Int. Ed. Engl., 2007, 46(33):6266-6270. doi: 10.1002/anie.v46:33 [48] BOSSI M, FOLLING J, BELOV V N, et al.. Multicolor far-field fluorescence nanoscopy through isolated detection of distinct molecular species[J]. J. Am. Chem. Soc., 2008, 8(8):2463-2468. http://cn.bing.com/academic/profile?id=526d08a731da80e2265c9712936088d9&encoded=0&v=paper_preview&mkt=zh-cn [49] GRIMM J B, SUNG A J, LEGANT W R, et al.. Carbofluoresceins and carborhodamines as scaffolds for high-contrast fluorogenic probes[J]. ACS Chem. Biol., 2013, 8(6):1303-1310. doi: 10.1021/cb4000822 [50] DENIZ E, TOMASULO M, CUSIDO J, et al.. Photoactivatable fluorophores for super-resolution imaging based on oxazine auxochromes[J]. The Journal of Physical Chemistry C, 2012, 116(10):6058-6068. doi: 10.1021/jp211796p [51] TIAN Z, LI A D, HU D. Super-resolution fluorescence nanoscopy applied to imaging core-shell photoswitching nanoparticles and their self-assemblies[J]. Chem. Commun.(Camb), 2011, 47(4):1258-1260. doi: 10.1039/C0CC03217D [52] ZHANG H, WANGC, JIANG T, et al.. Microtubule-targetable fluorescent probe:site-specific detection and super-resolution imaging of ultratrace tubulin in microtubules of living cancer cells[J]. Anal. Chem., 2015, 87(10):5216-5222. doi: 10.1021/acs.analchem.5b01089 [53] LI C, HU Z, ALDRED M P, et al.. Water-soluble polymeric photoswitching dyads impart super-resolution lysosome highlighters[J]. Macromolecules, 2014, 47(24):8594-8601. doi: 10.1021/ma501505w [54] BATES M, HUANG B, DEMPSEY G T, et al.. Multicolor Super-Resolution Imaging with Photo-Switchable Fluorescent Probes[J]. Science, 2007, 317:1749-1753. doi: 10.1126/science.1146598 [55] HUANG B, WANG W, BATES M, et al.. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy[J]. Science, 2008, 319:810-813. doi: 10.1126/science.1153529 [56] HEILEMANN M, VAN D L S, SCHUTTPELZ M, et al.. Subdiffraction-resolution fluorescence imaging with conventional fluorescent probes[J]. Angew Chem. Int. Ed. Engl., 2008, 47(33):6172-6176. doi: 10.1002/anie.v47:33 [57] GRAHAM T DEMPSEY, BATES M, WALTER E KOWTONIUK, et al.. Photoswitching mechanism of cyanine dyes[J]. J. Am. Chem. Soc., 2009, 131:18192-18193. doi: 10.1021/ja904588g [58] VAUGHAN J C, DEMPSEY G T, SUN E, et al.. Phosphine quenching of cyanine dyes as a versatile tool for fluorescence microscopy[J]. J. Am. Chem. Soc., 2013, 135(4):1197-2000. doi: 10.1021/ja3105279 [59] FU N, XIONG Y, SQUIER T C. Synthesis of a targeted biarsenical Cy3-Cy5 affinity probe for super-resolution fluorescence imaging[J]. J. Am. Chem. Soc., 2012, 134(45):18530-18533. doi: 10.1021/ja308503x [60] GUNSOLUS I L, HU D, MIHAI C, et al.. Facile method to stain the bacterial cell surface for super-resolution fluorescence microscopy[J]. Analyst, 2014, 139(12):3174-3178. doi: 10.1039/C4AN00574K [61] CHIEN M P, CARLINI A S, HU D, et al.. Enzyme-directed assembly of nanoparticles in tumors monitored by in vivo whole animal imaging and ex vivo super-resolution fluorescence imaging[J]. J. Am. Chem. Soc., 2013, 135(50):18710-18713. doi: 10.1021/ja408182p [62] HEILEMANN M, VAN D L S, MUKHERJEE A, et al.. Super-resolution imaging with small organic fluorophores[J]. Angew Chem. Int. Ed. Engl., 2009, 48(37):6903-6908. doi: 10.1002/anie.v48:37 [63] LUKINAVICIUS G, UMEZAWA K, OLIVIER N, et al.. A near-infrared fluorophore for live-cell super-resolution microscopy of cellular proteins[J]. Nat. Chem., 2013, 5(2):132-139. doi: 10.1038/nchem.1546 [64] UNO S N, KAMIYA M, YOSHIHARA T, et al.. A spontaneously blinking fluorophore based on intramolecular spirocyclization for live-cell super-resolution imaging[J]. Nat. Chem., 2014, 6(8):681-689. doi: 10.1038/nchem.2002 [65] SCHAFER P, VAN D L S, LEHMANN J, et al.. Methylene blue-and thiol-based oxygen depletion for super-resolution imaging[J]. Anal. Chem., 2013, 85(6):3393-3400. doi: 10.1021/ac400035k [66] LEE S F, VEROLET Q, FURSTENBERG A. Improved super-resolution microscopy with oxazine fluorophores in heavy water[J]. Angew Chem. Int. Ed. Engl., 2013, 52(34):8948-8951. doi: 10.1002/anie.201302341 [67] DERTINGER T, RYAN C, VOGEL R, et al.. Achieving increased resolution and more pixels with SOFI[J]. Optics Express, 2010, 18(18):18875-18885. doi: 10.1364/OE.18.018875 [68] GEISSBUEHLER S, DELLAGIACOMA C, LASSER T. Comparison between SOFI and STORM[J]. Optics Express, 2011, 2(3):408-420. doi: 10.1364/BOE.2.000408 [69] GEISSBUEHLER S, BOCCHIO N L, DELLAGIACOMA C, et al.. Mapping molecular statistics with balanced super-resolution optical fluctuation imaging(bSOFI)[J]. Optical Nanoscopy, 2012, 1:1-7. doi: 10.1186/2192-2853-1-1 [70] GEISSBUEHLER S, SHARIPOV A, GODINAT A, et al.. Live-cell multiplane three-dimensional super-resolution optical fluctuation imaging[J]. Nat. Commun., 2014, 5:5830. doi: 10.1038/ncomms6830 [71] WANG X H, CHEN D N, YU B, et al.. Deconvolution optimization in super-resolution optical fluctuation imaging based on cumulant standard deviation[J]. Acta Physica Sinica, 2016, 65:198701. [72] ZENG Z P, CHEN X Z, WANG H, et al.. Fast super-resolution imaging with ultra-high labeling density achieved by joint tagging super-resolution optical fluctuation imaging[J]. Sci. Rep., 2015, 5:8359. doi: 10.1038/srep08359 [73] HABUCHI S, ANDO R, DEDECKER P, et al.. Reversible single-molecule photoswitching in the GFP-like fluorescent protein Dronpa[J]. Proc. Natl. Acad. Sci. USA, 2005, 102(27):9511-9516. doi: 10.1073/pnas.0500489102 [74] DEDECKER P, MO G C, DERTINGER T, et al.. Widely accessible method for superresolution fluorescence imaging of living systems[J]. Proc. Natl. Acad. Sci. USA, 2012, 109(27):10909-10914. doi: 10.1073/pnas.1204917109 [75] ZHANG M, CHANG H, ZHANG Y, et al.. Rational design of true monomeric and bright photoactivatable fluorescent proteins[J]. Nat. Methods, 2012, 9(7):727-729. doi: 10.1038/nmeth.2021 [76] ZHANG X, CHEN X Z, ZENG Z P, et al.. Development of a reversibly switchable fluorescent protein for super-resolution optical fluctuation imaging(SOFI)[J]. ACS Nano, 2015, 9(3):2659-2667. doi: 10.1021/nn5064387 [77] DERTINGER T, HEILEMANN M, VOGEL R, et al.. Superresolution optical fluctuation imaging with organic dyes[J]. Angew Chem. Int. Ed. Engl., 2010, 49(49):9441-9443. doi: 10.1002/anie.201004138 [78] DAVID A. VANDEN B, WAI T Y, HU D H, et al.. Discrete intensity jumps and intramolecular electronic energy transfer in the spectroscopyof single conjugated polymer molecule[J]. Science, 1997, 277:1074-1077. doi: 10.1126/science.277.5329.1074 [79] BARBARA P F, GESQUIERE A J, PARK S J, et al.. Single-molecule spectroscopy of conjugated polymers[J]. Acc. Chem. Res., 005, 38:602-610. doi: 10.1021/ar040141w [80] WU C F, CHIU D T. Highly fluorescent semiconducting polymer dots for biology and medicine[J]. Angew Chem. Int. Ed. Engl., 2013, 52(11):3086-3109. doi: 10.1002/anie.201205133 [81] WU C F, SZYMANSKI C, CAIN Z, et al.. Conjugated polymer dots for multiphoton fluorescence imaging[J]. J. Am. Chem. Soc., 2007, 129:12904-12905. doi: 10.1021/ja074590d [82] WU C F, BARBARA B, SZYMANSKI C, et al.. Multicolor conjugated polymer dots for biological fluorescence imaging[J]. ACS Nano, 2008, 2(11):2415-2423. doi: 10.1021/nn800590n [83] CHEN X Z, LI R Q, LIU Z H, et al.. Small Photoblinking Semiconductor Polymer Dots for Fluorescence Nanoscopy[J]. Advanced Materials, 2017, 29(5). http://cn.bing.com/academic/profile?id=ee2f4456344a9b0f757c13a347e0a321&encoded=0&v=paper_preview&mkt=zh-cn [84] CHEN X Z, LIU Z H, LI R Q, et al.. Multicolor super-resolution fluorescence microscopy with blue and carmine small photoblinking polymer dots[J]. ACS Nano, 2017. http://cn.bing.com/academic/profile?id=ca7d3f868f8de12b0eda8aa67704e384&encoded=0&v=paper_preview&mkt=zh-cn -





 下载:
下载: